
|
Опрос
|
реклама
Быстрый переход
Учёные научились «консервировать» солнечное тепло на зиму — на открытие навела химия ДНК
17.02.2026 [14:18],
Геннадий Детинич
Простой нагрев воды в солнечном коллекторе на крыше не позволяет запасать тепло надолго. Было бы отлично нагреть теплоноситель летом, а расходовать зимой. Именно к этому стремятся разработчики систем накопления тепла на основе молекулярной тепловой инверсии, когда под действием света молекула впитывает энергию, а затем контролируемо её отдаёт. В этой области долго не было прорывов, но теперь наша ДНК подсказала верное направление. 
Пример солнечного коллектора для нагрева воды. Источник изображения: Kypros Исследователи из Университетов Калифорнии в Санта-Барбаре и в Лос-Анджелесе разработали производное 2-пиримидона — соединения, родственного тимину в составе ДНК. Идея заимствована из природного процесса: ультрафиолетовое излучение вызывает в ДНК повреждения, которые могут превращаться в высокоэнергетические изомеры Дьюара. И если у человека это может привести к онкологии, то в системе накопления тепла — это просто способ запасти энергию на длительный период. В организме такие повреждения восстанавливаются специальным ферментом, а в технических системах энергия высвобождается с помощью катализаторов. Синтезированная исследователями молекула поглощает ультрафиолетовый свет в диапазоне UV-A и UV-B (примерно 300–310 нм). Возникающий при этом изомер отличается высокой стабильностью — его период полураспада достигает 481 дня при комнатной температуре. Это позволяет хранить энергию месяцами без значительных потерь: накопить её жарким июлем и расходовать в январе. Жидкая при комнатной температуре молекула хорошо растворяется в воде, не требует токсичных органических растворителей и в экспериментах выдержала 20 циклов заряда/разряда с минимальной деградацией. Созданные до этого молекулярные теплоносители требовали токсичных растворителей и поэтому теряли плотность запасаемой энергии, тогда как жидкая природа новой молекулы позволяет использовать её в неразбавленном виде. Впрочем, она растворяется в воде, что делает её легко удаляемой, например, в случае протечек в домашних условиях. Процесс разрядки — выделения запасённого тепла — запускается добавлением кислотного катализатора. Тепло уходит в теплообменник и обогревает дом. Но пока в этом кроется минус: добавление катализатора разбавляет теплоноситель и снижает плотность накопления энергии. Учёным ещё предстоит решить эту проблему, чтобы катализатор оставался отделённым от основного объёма. Тем не менее полученные в опытах рекордные характеристики делают разработку выдающейся: плотность хранимой энергии достигает 1,65 МДж/кг, что почти вдвое превышает показатели литийионных аккумуляторов (менее 1 МДж/кг) и значительно превосходит предыдущие молекулярные материалы (норборнадиен — 0,97 МДж/кг, азоборинин — 0,65 МДж/кг). Это открывает перспективы для компактного сезонного хранения солнечного тепла, особенно для отопления зданий: жидкость может циркулировать через солнечные коллекторы на крыше, заряжаться, храниться в резервуарах в подвалах и по мере необходимости пропускаться через катализатор для передачи тепла в систему отопления или горячего водоснабжения. Предложенный подход позиционируется как экологичная альтернатива традиционному топливу для зимнего периода. Несмотря на впечатляющие результаты, технология имеет ограничения, препятствующие немедленному коммерческому внедрению. Молекула использует лишь около 5 % солнечного спектра (только узкий диапазон УФ), не реагируя на видимый свет и инфракрасное излучение, а квантовая эффективность превращения остаётся низкой (реагируют лишь несколько фотонов из каждых 100), что требует длительного облучения. Кроме того, применение кислотного катализатора усложняет систему и требует дополнительных шагов для его нейтрализации. Авторы подчёркивают необходимость дальнейших улучшений — расширения спектра поглощения и упрощения механизма разрядки, — чтобы сделать технологию практически применимой в реальных условиях. ИИ превзошёл эволюцию в синтезе важнейших элементов ДНК — генная терапия готова совершить новый виток
03.01.2026 [14:55],
Геннадий Детинич
Современная генетика всё чаще обращается к искусственному интеллекту как к инструменту не только анализа, но и создания новых биологических элементов. В новой работе учёные показали, что генеративные модели ИИ способны проектировать регуляторные участки ДНК — фрагменты, отвечающие за экспрессию генов. Это новый шаг: вместо поиска подходящих регуляторов в природе исследователи начали создавать их с нуля под конкретные задачи. 
Источник изображения: ИИ-генерация ChatGPT 5.2/3DNews В основе работы лежит метод DNA-Diffusion — алгоритм, заимствующий идеи из диффузионных моделей, широко применяемых в генерации изображений и текста. Модель обучалась на огромных массивах геномных данных и научилась предсказывать, какие последовательности ДНК будут эффективно включать или усиливать работу генов в определённых типах клеток. В результате ИИ смог сгенерировать тысячи вариантов синтетических регуляторов, многие из которых оказались функционально сильнее природных аналогов. Экспериментальная проверка показала, что созданные ИИ регуляторные элементы действительно работают в живых клетках. Более того, они продемонстрировали высокую точность: активировали гены именно в нужных клетках, минимизируя побочные эффекты. Особенно показательным стал пример с геном AXIN2 (важным для подавления лейкемии) — синтетические регуляторы управляли его активностью эффективнее природных защитных вариантов. Значение этой работы выходит далеко за рамки фундаментальной науки. Возможность целенаправленно конструировать регуляторные фрагменты ДНК открывает путь к более безопасной и точной генной терапии, созданию «умных» генетических лекарств и тонкой настройке клеточных функций. По сути, ИИ начинает выступать в роли инженера генома, расширяя границы того, что ранее считалось возможным только в ходе естественного эволюционного процесса. В США представили «вечную флешку» на базе ДНК — терабайты в капсуле размером с таблетку
03.12.2025 [19:08],
Геннадий Детинич
Американская биотехнологическая компания Atlas Data Storage представила революционный сервис для хранения данных на синтетической ДНК — Atlas Eon 100, который решает проблему сохранения колоссальных массивов данных. Предложенная разработчиком платформа позволит записывать цифровые архивы с надёжностью, недоступной современным электронным носителям, обещая сохранность информации на многие тысячи лет. 
Источник изображения: Atlas Data Storage Исследователи давно пытаются создать системы хранения цифровой информации на ДНК — этом чуде биологической эволюции, благодаря которому на Земле существует сама жизнь. У системы записи данных на ДНК есть один существенный недостаток — медленное время записи и чтения, поскольку все процессы представляют собой растянутые во времени химические реакции. Но главное преимущество ДНК — способность сохраняться тысячелетиями — покрывает все минусы этой технологии. Компания Atlas Data Storage представила сервис, а не устройство типа флешки или жёсткого диска для самостоятельного использования — до этого пока далеко. Она берётся перевести любые цифровые данные клиента в код для записи на синтетической ДНК, для чего вместо нулей и единиц используются четыре нуклеотида: аденин (A), цитозин (C), гуанин (G) и тимин (T). Затем компания синтезирует ДНК с записанной информацией, обезвоживает её и передаёт заказчику в специальной герметичной капсуле для вечного хранения.  Для чтения информации требуется секвенировать содержимое капсулы. Специальных ридеров для этого пока нет. Компания Atlas Data Storage использует в своей платформе стандартные коммерческие секвенсоры. Декодирование происходит в обычной офисной обстановке без каких-либо специальных условий. После секвенирования данные восстанавливаются в двоичном коде. Плотность хранения данных в системе Atlas Eon 100 в 1000 раз превышает плотность записи на магнитные ленты, а благодаря встроенным кодам коррекции ошибок надёжность восстановления данных достигает 99,99999999999 %. Точная ёмкость картриджа не раскрывается. По словам разработчика, она — масштаба терабайт. Цена вопроса тоже не афишируется. Сервис предложен корпорациям, государству, музейным фондам и состоятельным гражданам. Microsoft предсказала биологические угрозы «нулевого дня» из-за ИИ
03.10.2025 [18:16],
Павел Котов
Экспертам Microsoft при помощи искусственного интеллекта удалось обнаружить уязвимости «нулевого дня» в системах биологической безопасности, которые используются для предотвращения несанкционированного использования ДНК. 
Источник изображения: Braňo / unsplash.com Такие системы блокируют продажу и покупку генетических последовательностей, которые могут использоваться для создания смертельно опасных токсинов и патогенов. Группе исследователей под руководством старшего научного сотрудника Microsoft Эрика Хорвица (Eric Horvitz) удалось обойти эту защиту способом, ранее неизвестным специалистам в области безопасности. Хорвиц и его сотрудники подключили алгоритмы генеративного ИИ, предназначенные для получения новых форм белков — подобные программы уже помогают в открытии новых препаратов. Проблема в том, что они могут иметь двойное назначение, генерируя структуры не только полезных, но и опасных молекул. В Microsoft с 2023 года начали изучать потенциал систем ИИ двойного назначения, чтобы установить, способны ли подобные системы помочь биотеррористам в производстве вредоносных белков. Эксперты компании попытались преодолеть защиту ПО для обеспечения биологической безопасности. При производстве белков исследователи обычно заказывают у коммерческих поставщиков последовательности ДНК, которые впоследствии можно ввести в клетку. При помощи ПО поступающие заказы сравниваются с известными токсинами и патогенами — при близких совпадениях срабатывают оповещения. В разработке схемы атаки Microsoft применяла несколько моделей для генерации белков, в том числе свою собственную EvoDiff; исследователи поставили цель перепроектировать токсины таким образом, чтобы они могли пройти контроль ПО, но сохранить свои смертоносные свойства. 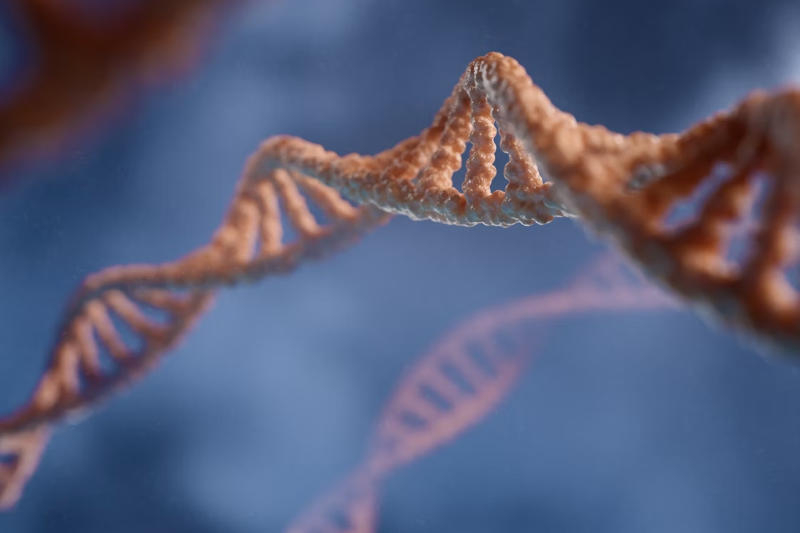
Источник изображения: digitale / unsplash.com Эксперимент проводился полностью в цифровой среде: в реальности они не создавали ни одного токсичного белка, чтобы избежать подозрений, что компания разрабатывает биологическое оружие. Microsoft также заявила, что перед публикацией материалов исследования оповестила о нём власти США и производителей ПО, хотя некоторые разработанные ИИ-молекулы всё равно обходят эту защиту. В компании Integrated DNA Technologies, крупном производителе ДНК-материалов, заявили, что закрытие этих уязвимостей — это не разовая работа, а «начало дальнейших испытаний», и сравнили сложившуюся ситуацию с «гонкой вооружений». Чтобы гарантировать, что результаты исследования не будут использованы не по назначению, исследователи из Microsoft не стали раскрывать подробностей своей работы и не сообщили, какие белки они просили ИИ переработать. Впрочем, есть мнение, что подобные точечные акции не помогут решить общую проблему, и лучшей защитой от злонамеренных действий было бы встроить средства защиты в сам ИИ — либо напрямую, либо посредством контроля над информацией, которую он выдаёт. Ещё более надёжным способом защиты является мониторинг самого синтеза генов на уровне производителя, потому что технологии ИИ становятся всё более распространёнными, и контролировать их труднее. «Этого джинна обратно в бутылку не загонишь. Если у тебя есть ресурсы, чтобы обманом попытаться заставить нас создать последовательность ДНК, ты, вероятно, сможешь и обучить большую языковую модель», — признают учёные. У мозга появился конкурент — ДНК-компьютер с невероятно доступным источником питания
02.10.2025 [22:17],
Геннадий Детинич
Практически все вдохновленные биологией разного рода молекулярные компьютеры сталкиваются с фундаментальной проблемой: отсутствием универсального источника энергии, подобного электричеству в компьютерах или АТФ в живых организмах. Это делает их одноразовыми, ограничивая выполнение задач до исчерпания «топлива». Учёные из США разработали ДНК-компьютер с невероятно доступным источником питания — это обычное тепло, которое можно найти везде. 
Источник изображения: ИИ-генерация Grok 3/3DNews Об открытии статьёй в журнале Nature сообщили исследователи Тяньци Сонг (Tianqi Song) и Лулу Цянь (Lulu Qian) из Калифорнийского технологического института (Caltech). Они разработали многослойные логические схемы и нейронные сети на основе ДНК, которые можно многократно перезаряжать простым циклом нагрева и охлаждения. Такой подход позволяет системам работать минимум 16 циклов без накопления отходов, открывая путь к автономным молекулярным вычислениям. У мозга появился конкурент: органика оказалась способной к сложным «раздумьям» на иных принципах без использования нервных тканей. Учёные нашли выход в том, что назвали кинетической ловушкой. Это стало механизмом перезарядки ДНК-компьютера. «Это как взведённая мышеловка», — поясняют исследователи. Она готова к действию от малейшего прикосновения. Нити ДНК специально спроектированы так, чтобы сгибаться в форму шпильки-невидимки для волос. При нагреве до 95 °C все нити разделяются и свободно плавают в растворе. При охлаждении они быстро формируют шпильки — одномолекулярные структуры. Эта шпилька остаётся заряженной — готовой к действию — до появления в растворе входных данных в виде молекул-катализаторов. Катализаторы высвобождают энергию в процессе распрямления молекул ДНК. В зависимости от входных данных, зашифрованных в молекулах-катализаторах, происходит вычислительный процесс — реакции молекул ДНК. Чтобы вернуть схему в первоначальное состояние, необходимо нагреть раствор и затем охладить его: молекулы ДНК снова распрямляются, а потом сворачиваются в форму шпилек. Такой простой цикл обеспечивает полную перезагрузку схемы за считаные минуты — без внесения химикатов и иной подпитки. Для эксперимента исследователи создали полноценную 100-битную нейронную сеть, способную классифицировать рукописные цифры «6» и «7» из базы данных MNIST. Система включала до 289 различных нитей ДНК в одной пробирке и использовала чередующиеся слои вентилей из «шпилек». Такая архитектура обеспечила бесперебойную передачу сигналов через несколько уровней, минимизируя помехи и повышая масштабируемость. Вычисления происходили без чипов или электричества, полагаясь исключительно на молекулярные взаимодействия, что наглядно продемонстрировало потенциал ДНК для решения сложных задач машинного обучения. 
Источник изображения: Nature 2025 После отработки алгоритма раствор с ДНК перезагружается: добавляются молекулы-ингибиторы для нейтрализации входных данных (молекул-катализаторов), затем следует нагрев для разделения нитей и охлаждение для нового формирования шпилек. Этот процесс был повторён для десяти тестовых изображений, подтвердив надёжность системы в обработке разнообразных данных. Отсутствие накопления отходов и стабильность на протяжении 16 циклов подчеркнули практическую жизнеспособность подхода и преодоление прежних ограничений ДНК-вычислений, где логические элементы быстро деградировали из-за побочных продуктов. Прорыв радикально расширяет понятие компьютера, показывая, что устойчивые вычисления возможны без проводов или кремния — достаточно простых изменений температуры. Он закладывает основу для автономных химических систем с вычислениями и обучением без контроля, во многом имитируя эволюцию жизни. Это также обостряет вопросы о природе интеллекта, противопоставляя наш эволюционировавший мозг «мышлению» в молекулярных нейронных сетях. Китайские учёные запихнули ДНК в кассету и создали накопитель нового поколения ёмкостью до 36 Пбайт
13.09.2025 [16:22],
Павел Котов
С постоянным расширением цифровизации вопрос хранения данных стоит всё более остро: ресурсы жёстких дисков и других носителей когда-нибудь себя исчерпают, потому что человек генерирует информацию быстрее, чем производит носители для её хранения. Одним из решений проблемы может стать то, что уже создала природа — дезоксирибонуклеиновая кислота или ДНК, которую учёные китайского Южного технологического университета поместили в кассеты для хранения данных. 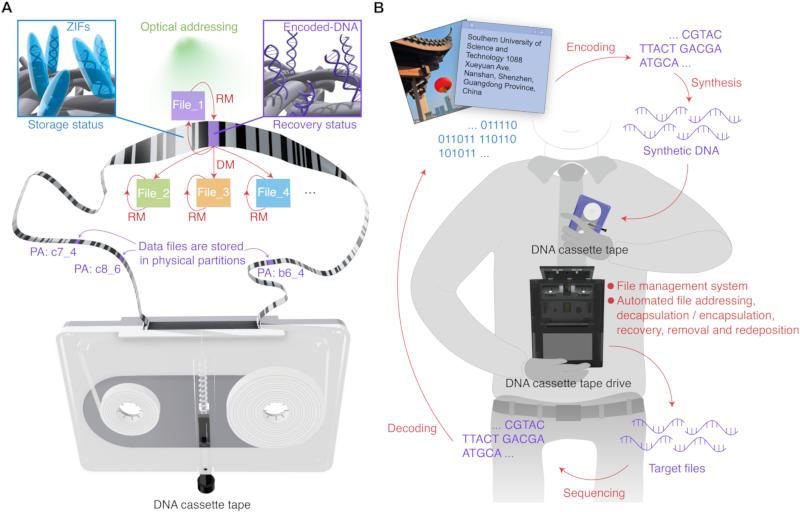
Источник изображения: science.org ДНК представляется идеальным решением для хранения данных, обладая такими свойствами как компактность, высокая плотность и долговечность. Она позволяет записывать огромные объёмы информации на носители микроскопических размеров и хранить их тысячи лет без подключения к электропитанию. Теоретически, в одну ДНК-клетку человека можно записать около 3,2 Гбайт данных — это эквивалентно примерно 6000 книг, 1000 музыкальных композиций или двум фильмам. Учёным давно известно о потенциале ДНК как средства хранения данных, и теперь перед ними стоит задача построить реальную жизнеспособную систему, которой можно было бы пользоваться. Китайские учёные разместили ДНК на носителе, напоминающем аудиокассету — такие в восьмидесятые годы прошлого века использовались в домашних магнитофонах и автомобильных магнитолах. Исследователи изготовили ленту из полиэстера и нейлона, после чего нанесли на неё нечто вроде штрихкода — несколько миллионов крошечных секций, как папки на компьютере. Такая схема позволяет точно определять место хранения данных, что важно, потому что аспект доступа к информации оставался одной из проблем при предыдущих разработках методов хранения ДНК. При записи файла цифровая последовательность данных преобразуется в последовательность ДНК — в качестве кода используются четыре основных строительных блока: A (аденин), G (гуанин), C (цитозин) и T (тимин), которые выступают в качестве аналога двоичному коду из нулей и единиц в компьютерах. Ленту учёные покрыли кристаллическим защитным слоем, препятствующим разрушению связей ДНК. И подтвердили работоспособность системы — показали преобразование цифрового изображения в формат ДНК и успешно считали данные с ленты.  Исследовательская группа добилась впечатляющей плотности хранения: до 28,6 миллиграмма ДНК на километр ленты и возможности обрабатывать до 1570 разделов в секунду. Чтобы представить объём: традиционная кассетная лента могла вместить примерно 12 песен на каждую сторону, тогда как 100 метров ДНК-кассеты способны сохранить более 3 миллиардов «песен» по 10 мегабайт каждая. «ДНК-кассета воплощает стратегию быстрого, компактного и масштабного хранения данных на основе ДНК — как „холодного“ (используемого редко), так и „горячего“ (предоставляемого по необходимости)», — пишут авторы исследования. Эта технология может стать масштабируемым решением для центров обработки данных, испытывающих нехватку пространства и энергоресурсов, и предложить замену громоздким системам хранения данных. Учёные придумали хранить данные в пластиковом аналоге ДНК — это будет плотно и надёжно
20.05.2025 [13:58],
Геннадий Детинич
По разным оценкам, объём цифровых данных в мире превысил 175 зеттабайт. Страшно представить, сколько это будет весить при записи на жёсткие диски. А ведь объём информации продолжает расти по экспоненте. Даже при хранении информации в ДНК, запись такого объёма данных потребует около одной тонны биологического материала. К счастью, этот способ записи значительно опережает другие по соотношению объёма данных к массе, и именно это привлекает внимание учёных. 
Источник изображения: ИИ-генерация Grok 3/3DNews Разработке технологий записи информации в ДНК посвящено множество научных работ. Природа за миллиарды лет эволюции отработала механизм передачи информации о наследственных свойствах и особенностях биологических организмов из поколения в поколение. Этот метод гарантирует высокую точность, надёжность и избыточность — всё это крайне важно для длительного хранения данных. ДНК могут сохраняться без повреждений десятки и даже сотни лет, а с учётом избыточности — десятки тысяч лет и более. Исследователи из Техасского университета в Остине (University of Texas at Austin) развили идею организации ДНК до записи данных в комбинации искусственных молекул. Они создали молекулы из пластика, которые, подобно нуклеотидам в ДНК, собираются в заданные последовательности. Такой подход может упростить создание практичного оборудования для записи и чтения псевдо-ДНК. Современные устройства для секвенирования ДНК крайне сложны, дороги и слабо подвержены тенденции к удешевлению и упрощению. В случае с искусственным аналогом всё может оказаться гораздо проще. Молекулы для кодирования и последующего прочтения информации можно будет подбирать более гибко. Учёные подобрали четыре различных пластиковых молекулы и зашифровали с их помощью длинную последовательность сложного шифровального ключа. Затем этот ключ был успешно прочитан. Для чтения таких записей в настоящее время используется лабораторный масс-спектрометр, который в процессе анализа молекул уничтожает носитель. Масс-спектрометр — это также сложное и дорогостоящее оборудование, которое пока не позволяет массово перейти на запись данных в псевдо-ДНК. Однако проведённая работа демонстрирует, что такое в принципе возможно. Создание большего набора молекул — шести, восьми и более с уникальными кодами — за счёт увеличения разрядности позволит ещё больше повысить плотность молекулярной записи. ИИ помог разработать противоядия от смертельных ядов змей
15.01.2025 [21:59],
Геннадий Детинич
Прогнозирование в сворачивании белков назвали одним из прорывов искусственного интеллекта в 2024 году. Словно подтверждая это, учёные сообщили сегодня о разработке с помощью ИИ противоядий от самых смертоносных змеиных ядов мамб, морских и гремучих змей. Глубокое машинное обучение в режиме диалога с исследователями помогло создать белки, защищающие организм от смертельных токсинов, что было подтверждено проверками на мышах. И это только начало. 
Источник изображения: Kate Zvorykina/Ella Maru Studio Сегодня противоядия создаются сложным и дорогостоящим методом иммунизации животных. Животным, например лошадям, вводят яд змей и ждут появления антител. Затем антитела извлекаются из забранной крови на центрифуге, что ведёт к образованию сыворотки — фактически лекарства. На всё это уходят недели и месяцы. Машинное обучение обещает радикально сократить время синтеза белков для нейтрализации токсинов. И это будет, конечно же, дешевле и безопаснее для пострадавших. Исследование провёл медицинский факультет Вашингтонского университета (UW Medicine) в сотрудничестве с коллегами из Медицинского института по разработке белков Калифорнийского университета и Технического университета Дании. Забегая вперёд, отметим, что университет подал заявку на получение патента на разработанное с помощью ИИ противоядие. Проблема с укусами змей ежегодно затрагивает свыше 2 млн человек. По данным Всемирной организации здравоохранения, более 100 000 из них умирают, а 300 000 страдают от серьёзных осложнений и даже становятся инвалидами в результате деформации конечностей, ампутации или других последствий. Страны Африки к югу от Сахары, Южная Азия, Папуа-Новая Гвинея и Латинская Америка входят в число мест, где укусы ядовитых змей представляют наибольшую проблему для общественного здравоохранения. Исследовательская группа сосредоточила своё внимание на поиске способов нейтрализации яда, полученного от определенных элапидов. Элапиды — это большая группа ядовитых змей, среди которых кобры и мамбы, обитающие в тропиках и субтропиках. Яд этой группы включает так называемый токсин трёх пальцев (three-finger toxin, 3FTx). Это химическое вещество повреждает ткани организма, убивая клетки, а также прерывает сигналы между нервами и мышцами, вызывая паралич и смерть. ИИ помог создать новые белки, которые вступали в соединения с токсинами и связывали их, не допуская к рецепторам клеток живого организма. Взаимодействуя с программой, учёные получали инструкции по синтезу термостабильных белков с высокой способностью связываться с токсинами. Практически синтезированные белки почти полностью соответствовали на атомарном уровне компьютерному дизайну с глубоким обучением. В лабораторных условиях разработанные белки эффективно нейтрализовали все три подсемейства тестируемых трёхпальцевых токсинов. При введении мышам разработанные белки защищали животных от того, что могло быть смертельным воздействием на них. Разработанные белки обладают множеством преимуществ. Их можно производить с неизменным качеством с помощью технологий рекомбинантной ДНК, а не путём иммунизации животных. Кроме того, новые белки, разработанные против змеиных токсинов, имеют небольшие размеры по сравнению с антителами. Их меньший размер может обеспечить большее проникновение в ткани для быстрого противодействия токсинам и уменьшения повреждений. В дополнение к открытию новых путей разработки противоядий, исследователи полагают, что методы компьютерного проектирования могут быть использованы для разработки других лекарств. Такие методы также могут быть использованы для поиска лекарств от так называемых пренебрегаемых заболеваний, которые поражают беднейшие страны в зоне тропиков, на что обычно никогда нет денег. Учёные построили из ДНК нанороботов с клешнями для отлова вирусов
28.11.2024 [12:49],
Геннадий Детинич
В Иллинойсском университете в Урбане-Шампейне (University of Illinois at Urbana-Champaign) создали NanoGripper — нечто вроде четырёхпалой руки из цельного фрагмента ДНК. Пальцы этой «руки» автоматически сжимаются вокруг вируса, реагируя на его молекулярный состав. После этого действие вируса блокируется — он не может проникнуть в клетку и заразить её. Кроме того, NanoGripper способен доставлять лекарства к клеткам-мишеням, что даёт надежду на его применение в борьбе с раком. 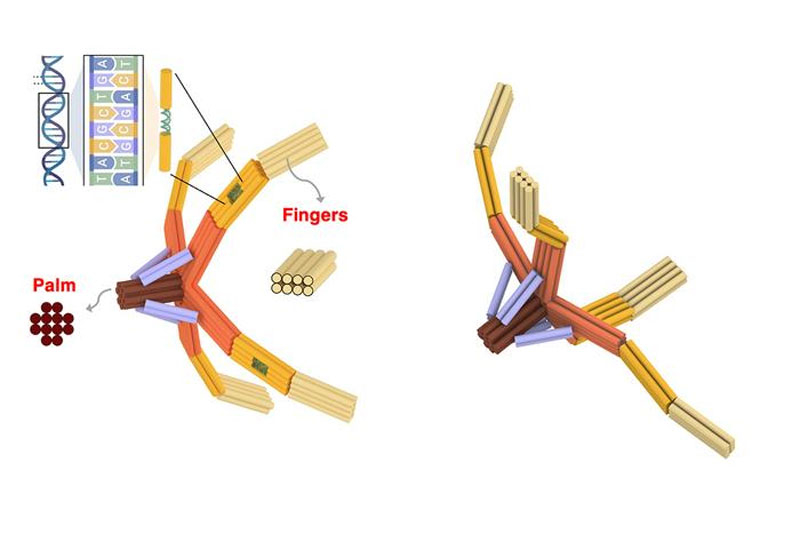
Источник изображений: University of Illinois at Urbana-Champaign По словам учёных, это первая работа такого рода, позволяющая из одной неразрывной нити ДНК создать захват нанометрового масштаба. Нить многократно сгибают, чтобы воспроизвести четырёхпалый захват с тремя подвижными суставами на каждом «пальце». NanoGripper был протестирован на захвате вируса COVID-19: чувствительные элементы суставов захвата были настроены на обнаружение спайковых белков вируса. Как только вирус попадал в зону действия захвата, он тут же обхватывался. Чужеродный элемент, закреплённый на вирусе, не давал ему проникнуть в клетку. В сочетании с диагностической системой это позволяло легко идентифицировать патоген. Зафиксированный и обездвиженный вирус становился мишенью для флуоресцентных молекул, что выдавало его присутствие. Таким образом, NanoGripper открывает возможность подсчёта вирусов в биоматериале, обеспечивая сверхточную диагностику. Предложенное решение не поможет вылечить уже заразившегося человека, но может использоваться в качестве профилактической меры. Например, в виде назального спрея, создающего защитный барьер против вирусов. Достаточно будет закапать нос, и своеобразные «капканы» для вирусных частиц будут готовы. 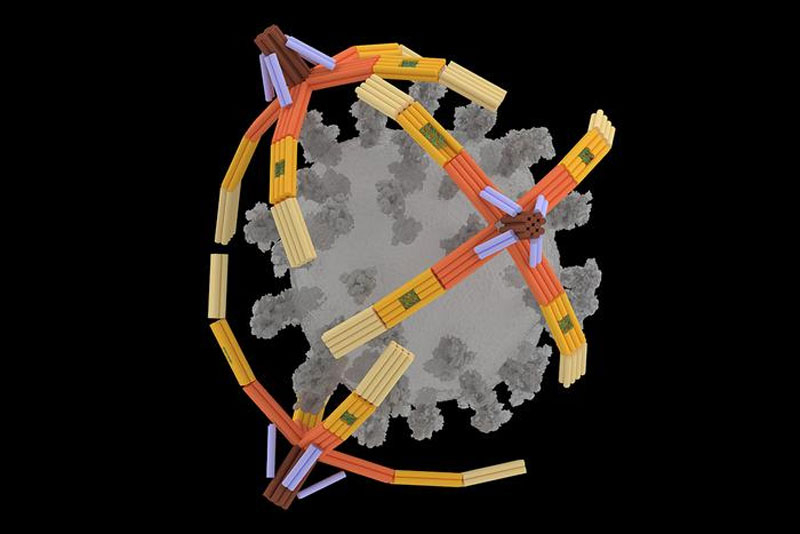 Как отмечают исследователи в своей работе, опубликованной в журнале *Science Robotics*, открытие имеет более широкий потенциал, чем заявлено в статье. NanoGripper может использоваться для доставки лекарств от рака непосредственно к клеткам, настройки на другие вирусы, такие как ВИЧ или гепатит, а также для диагностики. «Мы стремились создать робота из мягкого материала наноразмерного масштаба с невиданными ранее функциями захвата, который мог бы взаимодействовать с клетками, вирусами и другими молекулами для биомедицинских применений, — поясняют учёные. — Мы используем ДНК из-за её уникальных структурных свойств: прочности, гибкости и программируемости. Даже для области ДНК-оригами это новаторство с точки зрения принципа проектирования. Мы сгибаем одну длинную нить ДНК взад и вперёд, чтобы за один шаг получить все элементы, как статичные, так и подвижные». Разработана технология записи данных в существующую ДНК
26.10.2024 [16:39],
Павел Котов
Китайские учёные разработали новый метод хранения данных в ДНК, способный произвести революцию в этой узкой области. Группа исследователей Пекинского университета и трёх других научных учреждений опубликовала работу, посвящённую применению метилирования ДНК для выборочной мутации «эпи-битов» на уже существующих цепочках ДНК. Это значительно ускоряет процесс записи данных, но применять технологию на практике пока рано. 
Источник изображения: MV-Fotos / pixabay.com Запись информации в ДНК позволяет добиться невероятно высокой плотности данных — до 215 Пбайт на 1 грамм, но процессы записи и чтения пока и очень дороги, и очень медленны. Традиционно размещение данных в ДНК означает создание последовательностей с нуля, а китайские учёные предлагают записывать информацию в уже существующие нити, что в теории поможет сэкономить время и средства. Метод «эпи-битов» основан на естественном процессе, который называется «метилированием ДНК» — он имитирует эволюцию, которую претерпевают нити ДНК в течение жизни. Учёные создали из нуклеиновых кислот 700 «подвижных типов» ДНК. Этот метод может реализовываться вручную или автоматически: в ходе тестирования исследователи сначала напечатали, а затем вызвали изображения размером 18 833 бита и 252 504 бита (31,5 кбайт) в автоматическом режиме со скоростью 350 битов за реакцию. Для записи и хранения используется система штрих-кодов, помогающая отметить, где находятся фрагменты данных, чтобы их можно было извлечь с заданным уровнем скорости и точности. Запись информации в ДНК вручную — относительно несложный процесс даже для неспециалистов: 60 добровольцев без опыта работы в биолаборатории при помощи сервиса хранения данных iDNAdrive вручную закодировали 5000 битов текстовых данных. Предложенный китайскими учёными метод хранения данных в ДНК использует сильные стороны этой технологии — высокие плотность и стабильность — и добавляет к ним программируемость и масштабируемость. Но применять её на практике ещё рано: сейчас запись информации производится на скорости около 40 бит/с — примерно в 30 млн раз медленнее, чем на традиционный жёсткий диск. Зато стоимость оказалась примерно в десять раз ниже, чем создание последовательности с нуля — достаточно купить условные «ручку и чернила». На рынке цены пока заоблачные — французский стартап Biomemory взимает €1000 за запись 1 кбайт на карту памяти с ДНК. Учёные создали основу для будущих ДНК-компьютеров, которые одновременно хранят и обрабатывают данные
17.09.2024 [16:04],
Геннадий Детинич
Запись информации в ДНК обещает кардинально повысить плотность цифровых архивов, а способность этих молекул воспроизводить последовательности нуклеотидных оснований сравнима с редактированием и исполнением кода. До недавних пор учёным удавалось либо одно, либо другое, что далеко от идеала — создания биокомпьютеров для одновременного хранения и обработки информации. Учёные из США утверждают, что у них появилось решение. 
Источник изображения: Pixabay По словам исследователей из Университета Северной Каролины (NC) и Университета Джонса Хопкинса (Johns Hopkins University), они создали буквально предшественника всех ДНК-компьютеров будущего — систему, которая обеспечивает полный набор вычислительных функций с использованием цепочек нуклеиновых кислот, таких как хранение, считывание, стирание, перемещение и перезапись данных, а также управление этими функциями, как это делает обычный программируемый компьютер. «Считалось, что, хотя хранение данных в ДНК может быть полезным для долгосрочного хранения информации, было бы трудно или невозможно разработать ДНК-технологию, которая охватывала бы весь спектр операций, присущих традиционным электронным устройствам, — поясняют авторы работы. — Мы продемонстрировали, что эти технологии, основанные на ДНК, жизнеспособны, потому что мы их создали». В основе разработки лежит технология упорядоченного или даже иерархического распределения ДНК, тогда как обычно учёные работали с ДНК, свободно плавающими в растворах. Для этого учёные создали разветвлённую «волокнистую» структуру из такого полимера, как дендриколлоид диаметром 50 мкм. ДНК как бы вплетались в древовидную структуру нитей полимера, что позволяло, например, упростить стирание и перезапись заданных участков подобно работе с жёстким диском. При этом чтение не разрушало информацию (ДНК), так как она извлекалась из основы с помощью воспроизведения нужных участков в РНК — естественной функции, миллиарды лет присущей механизму дупликации с использованием ДНК. Одним из важнейших открытий стал найденный учёными способ отличать ДНК от основания (от волокон, в которые вплетены эти молекулы). Далее учёные показали, что с этими данными (с нуклеотидными основаниями) можно производить вычисления, как на обычном компьютере. Искусственное старение образцов показало, что при температуре 4 °C информация может сохраняться до 6000 лет, а при заморозке до -18 °C — до 2 млн лет. В одном кубическом сантиметре предложенная основа — дендриколлоид — сможет хранить до 10 Пбайт данных. Это хорошая заявка на расширение ёмкостей для длительного хранения архивов, которые смогут пережить не одну цивилизацию на Земле. Учёные облачили ДНК в искусственный янтарь — получилось сверхплотное и долговечное хранилище данных
14.06.2024 [17:53],
Павел Котов
ДНК — значительно более плотный носитель данных, чем что-либо, что создали люди, но проблема в том, что этот носитель крайне хрупкий. Для её решения учёные решили воспользоваться ещё одним достижением природы и создали искусственный янтарь, способный защитить хранящиеся в ДНК данные в течение длительного времени. 
Источник изображений: news.mit.edu Один грамм ДНК способен хранить до 215 Пбайт (215 млн гигабайт) данных — объёма с обувную коробку хватило бы на весь интернет. Это побудило учёных начать экспериментировать с записью данных в ДНК и их последующим считыванием, но необходимо обеспечить и их сохранность. Поэтому исследователи Массачусетского технологического института (США) создали своего рода искусственный янтарь, способный обеспечить защиту ДНК в течение длительного времени, а также последующее считывание данных. Это термореактивный материал — при нагревании он становится стекловидным твёрдым веществом, а при необходимости разрушается под воздействием химических соединений. 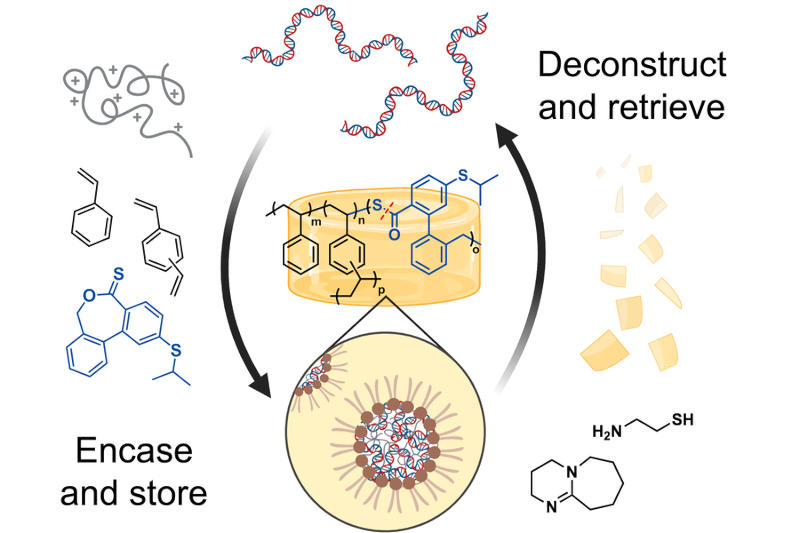 Учёные создали смесь мономеров, которые формируют сферические комплексы ДНК внутри и имеют водоотталкивающий слой снаружи — он нужен, потому что влага может повредить ДНК. Смесь нагревается и образует стекловидный блок, а ДНК с сохранёнными данными остаётся внутри. Когда возникает потребность считать эти данные, искусственный янтарь подвергается воздействию цистеамина — вещества, который его разрушает. После этого применяется детергент (моющее средство) под названием SDS, который отделяет ДНК, не повреждая её. Технологии присвоили название T-REX (Thermoset-REinforced Xeropreservation). На испытаниях учёные успешно применили технологию T-REX для хранения последовательностей ДНК различной длины при температурах до 75 °C. На тестовые последовательности они записали изданную Авраамом Линкольном «Прокламацию об освобождении рабов», логотип Массачусетского технологического института и музыкальную тему из фильма «Парк юрского периода» (Jurassic Park). Когда ДНК извлекли и секвенировали, ошибок обнаружено не было. Сейчас на процесс T-REX требуются несколько часов, и учёные говорят, что его можно оптимизировать. Едва ли накопители на основе ДНК когда-нибудь появятся в потребительских компьютерах, но для долгосрочного архивного хранения данных она может оказаться полезной. |